LABEL: Automatisches Labelling der Anatomien in großskaligen medizinischen Bilddatensätzen durch sElbst-überwachtes multimodales Lernen (DFG)
Die Analyse medizinischer Bilddaten hat in der Vergangenheit dank der neuartigen Deep-Learning-Techniken große Fortschritte gemacht. Für eine erfolgreiche Etablierung im Gesundheitswesen fehlen jedoch noch große Datensätze, die einen umfassenden Querschnitt der Bevölkerung repräsentieren und eine zuverlässige Erkennung von normaler Anatomie und Anomalien durch maschinelles Lernen ermöglichen.
Das Ziel des LABEL-Projekts ist die Entwicklung robuster und effizienter Algorithmen für die automatische Segmentierung der großen, umfassenden Bevölkerungsstudie NAKO, die mehr als 30.000 Ganzkörper-MRT-Scans enthält.
Im Mittelpunkt des LABEL-Projekts steht die Weiterentwicklung von Methoden zur automatischen Segmentierung von MR-Bildern mit möglichst wenigen annotierten Beispielen. Zu diesem Zweck werden Techniken des selbstüberwachten Pre-Trainings, des multimodalen Transferlernens und der lernbasierten Bildregistrierung kombiniert und zu neuartigen Algorithmen erweitert.
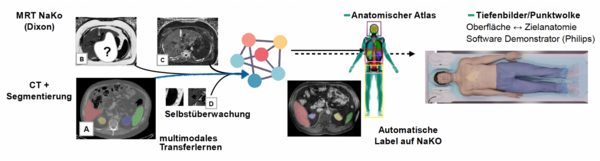
Darüber hinaus wird ein geometrischer 3D-Atlas der inneren Anatomie erstellt, der auf einem automatisch beschrifteten, groß angelegten NAKO-Datensatz basiert. Dieser Atlas wird in Verbindung mit Techniken des geometrischen Deep Learning die Entwicklung von Algorithmen zur präzisen Lokalisierung innerer Organe auf der Körperoberfläche ermöglichen. Die Integration der automatisierten Anatomie-Lokalisierung in den MRT-Aufnahme-Workflow wird den gesamten Scan-Prozess verkürzen und einen erheblichen wirtschaftlichen Vorteil bieten.
Im Rahmen des LABEL-Projekts werdendrei Partner - die Universität zu Lübeck (IMI), das Fraunhofer-Institut (MEVIS) und Phillips - bei der Entwicklung neuartiger Deep-Learning-Methoden zusammenarbeiten.
Das Projekt wird von dem DFG mit 426.360€ gefördert.
Ausgewählte Publikationen:
Blendowski, M., Nickisch, H., Heinrich, M.P. How to Learn from Unlabeled Volume Data: Self-supervised 3D Context Feature Learning. Medical Image Computing and Computer Assisted Intervention - MICCAI, 2019.
Blendowski, M., Bouteldja, N., Heinrich, M.P. Multimodal 3D medical image registration guided by shape encoder–decoder networks. Int J CARS 15, 269–276, 2020.
Hansen L, Heinrich M.P. GraphRegNet: Deep Graph Regularisation Networks on Sparse Keypoints for Dense Registration of 3D Lung CTs. IEEE Trans Med Imaging, 2021.
Projektteam:

- Forschung
- KI und Deep Learning in der Medizin
- Medizinische Bildverarbeitung und VR-Simulation
- Integration und Nutzbarmachung von medizinischen Daten
- Sensordatenanalyse für assistive Gesundheitstechnologien
- AG Medical Image Computing and Artificial Intelligence
- AG Medical Data Science
- AG Medical Deep Learning
- AG Medical Data Engineering
- Nachwuchsgruppe Diagnostik und Erforschung von Bewegungsstörungen
Ansprechpartner
Mattias Heinrich

Professor
Gebäude MFC2, 4.OG
mattias.heinrich(at)uni-luebeck.de
+49 451 3101 5602
