MDMLA: Multi-task Lernen mit tiefen Faltungsnetzwerken für großskalige Analyse von multimodalen biomedizinischen Bildern (BMBF)
Es ist gängige wissenschaftliche Vorgehensweise bei der Untersuchung physikalischer Objekte ist die Anwendung verschiedener, komplementärer experimenteller Methoden welche sich in ihrer räumlichen und zeitlichen Auflösung, Dimensionalität und modalitätsspezifischen Beschaffenheit unterscheiden und zu einer großen Menge an auszuwertenden Bilddaten führt.
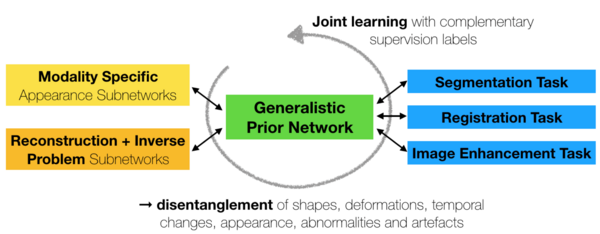
In diesem interdisziplinären Projekt soll ein generalistisches Deep Learning Framework entwickelt und implementiert werden, welches eine effiziente, modalitäts- und domänenübergreifende Verarbeitung und Analyse von multimodalen und longitudinalen biomedizinischen Daten ermöglicht. In enger Zusammenarbeit von Experten aus der Anwender- sowie Machine-Learning-Domäne (DESY, HZG, Syntellix, IMT, IMI) werden neue Multitask Deep Learning Methoden entwickelt welche die unterschiedlichen komplementären Aufgaben ganzheitlich miteinander kombinieren und damit Wissen über die einzelnen Analyseaufgaben hinweg zu transferieren. Anschließend werden generische Strukturen innerhalb der entwickelten Netzwerkarchitekturen identifiziert um ein vereinheitlichtes, datengesteuertes Framework zu entwerfen welches auf eine Vielzahl unterschiedlicher Domänen, Modalitäten und Aufgaben anwendbar ist.
Die entwickelten Methoden und Werkzeuge werden auf eine Vielzahl von longitudinalen und multimodalen Datensätzen aus vorklinischen Studien und klinischen Anwendungen, u.A. für die Untersuchung von Knochenimplantaten angewandt. Die Umsetzung der Methoden auf einer geeigneten Hochleistung-Rechner-Plattform wird zu einem langfristig nutzbaren Dienst führen, der einer Vielzahl von wissenschaftlichen Nutzern zur Verfügung stehen wird.
Am IMI sollen im Rahmen dieses Projektes, u.A. neue Ansätze für Deep Learning basierte Bildregistrierung und Multi-Task Lernen entwickelt werden. Hierzu planen wir insbesondere auch die Merkmalsextraktion und die geometrischen Modellierung stärker zu entkoppeln um die bestmöglichen Lösungsansätze für eine Vielzahl von Aufgaben (Rekonstruktion, Registrierung, Segmentierung) und Modalitäten (Labor-Computertomographie (labCT), Synchrotronstrahlungs-Mikro-CT (SRμCT), Magnetresonanztomographie (MRI), Kleinwinkel-Röntgenstreuung (SAXS) und Histologie) zu ermöglichen.
BMBF Projekt Förderung (2020-2023) 1‘170‘157€ (507‘163€ UzL)
Ausgewählte Publikationen
- Heinrich M.P., Hansen L.
Unsupervised learning of multimodal image registration using domain adaptation with projected Earth Move's discrepancies
Medical Imaging with Deep Learning (MIDL 2020, Montreal, short-paper)

- Forschung
- KI und Deep Learning in der Medizin
- Medizinische Bildverarbeitung und VR-Simulation
- Integration und Nutzbarmachung von medizinischen Daten
- Sensordatenanalyse für assistive Gesundheitstechnologien
- AG Medical Image Computing and Artificial Intelligence
- AG Medical Data Science
- AG Medical Deep Learning
- Nachwuchsgruppe Diagnostik und Erforschung von Bewegungsstörungen
- Ehemalige AG Medical Data Engineering
Ansprechpartner
Mattias Heinrich

Professor
Gebäude MFC2, 4.OG
mattias.heinrich(at)uni-luebeck.de
+49 451 3101 5602
