Artefaktreduzierte Rekonstruktion von 4D-CT-Bilddatensätzen aus 3D-CT-Datensegmenten
Problemstellung
Moderne CT-Scanner können mehrere Schichten gleichzeitig aufnehmen. Dennoch ist das in einer Rotation des Scanners aufgenommene Volumen häufig zu klein, um Organbewegungen erfassen zu können. Aber gerade in Gebieten wie der Radioonkologie ist es wichtig, auch Organbewegungen, die z.B. durch die Atmung entstehen, erfassen und vermessen zu können. Aus diesem Grund wurden in den USA Tumorpatienten mehrfach mit einem CT-Scanner untersucht und einzelne 3D-Datensegmente gewonnen. Aus diesen lassen sich 4D-CT Datensätze erzeugen, die zur weitergehenden Analyse der atmungsbedingten Organbewegungen verwendet werden sollen.
Methode
Ein moderner Mehrzeilen-CT wurde eingesetzt, um bei freier Atmung der Patienten einen bestimmten Körperabschnitt zu scannen (= Datensegment). Die jeweilige Atemlage wurde mit einem Spirometer gemessen, so dass mehrere Datensegment zu unterschiedlichen Phasen des Atemzyklus aufgenommen wurden konnten. Durch Verschieben des CT-Tisches konnten so Datensegmente für den gesamten Thoraxraum gewonnen werden.
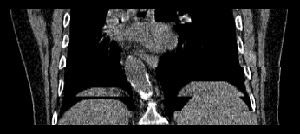
Zur Rekonstruktion eines 4D-CT Datensatzes aus den einzelnen Datensegmenten sind die gemessenen 3D-CT Datensegmente aufzubereiten. Hierfür werden Datensegmente an den einzelnen Tischpositionen benötigt, die zum selben Zeitpunkt des Atemzyklus, d.h. mit einem bestimmten Lungenvolumen, aufgenommen worden sind. In werden zur Erzeugung von 4D-Datensätzen jeweils die Datensegmente ausgewählt, die zu einem Lungenvolumen korrespondieren, das zu dem betrachteten Volumen am ähnlichsten ist (Abb. 1). Dies entspricht einer Nearest-Neighbour-Interpolation. Bei Einsatz dieser Methode treten jedoch Artefakte auf, die in Abb. 1 insbesondere im Bereich des Zwerchfells deutlich zu erkennen sind.
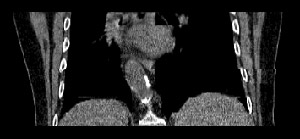
Zur Reduktion dieser Artefakte wurde ein Interpolationsverfahren entwickelt, das nicht-lineare Registrierungsalgorithmen verwendet. Hierbei wird eine nicht-lineare Registrierungsmethode eingesetzt, die auf dem "optischen Fluss" basiert. Angewandt auf zwei Datensegmente erhält man hierdurch ein vektorielles Verschiebungsfeld, das die Verschiebung der einzelnen Voxel zwischen den betrachteten Lungenvolumina approximativ beschreibt. Entlang des 3D-Verschiebungsfeldes können nun die Datensegmente zu den gewünschten Lungenvolumina interpoliert werden. Die so interpolierten 4D-Daten (Abb. 2) enthalten deutlich weniger Artefakte als die mit der Nearest-Neighbour-Strategie interpolierten (vgl. Abb. 1).
Ausgewählte Publikationen
- Jan Ehrhardt, René Werner, Thorsten Frenzel, Dennis Säring, Wei Lu, Daniel Low, Heinz Handels: Optical Flow based Method for Improved Reconstruction of 4D CT Data Sets Acquired During Free Breathing.
Medical Physics, 34, 2, 711-721, 2007. - René Werner, Jan Ehrhardt, Thorsten Frenzel, Dennis Säring, Wei Lu, Daniel Low, Heinz Handels: Motion Artifact Reducing Reconstruction of 4D CT Image Data for the Analysis of Respiratory Dynamics.
Methods of Information in Medicine, 46, 254-260, 2007. - Heinz Handels, René Werner, Thorsten Frenzel, Dennis Säring, Wei Lu, Daniel Low, Jan Ehrhardt: Generation of 4D CT Image Data and Analysis of Lung Tumour Mobility During the Breathing Cycle.
Stud Health Technol Inform, 124, 977-982, 2006.
Projektteam
Dr. Jan Ehrhardt
Dipl.-Inf. Dipl.-Phys. René Werner
Prof. Dr. Heinz Handels
Kooperationspartner
Prof. Daniel Low and Dr. Wei Lu
Mallinckrodt Institute of Radiology
Washington University
St. Louis, USA
Dr. med. Dr. rer. nat. Thorsten Frenzel
Klinik und Poliklinik für Strahlentherapie und Radioonkologie
Universitätsklinikum Hamburg-Eppendorf

- Forschung
- KI und Deep Learning in der Medizin
- Medizinische Bildverarbeitung und VR-Simulation
- Integration und Nutzbarmachung von medizinischen Daten
- Sensordatenanalyse für assistive Gesundheitstechnologien
- AG Medical Image Computing and Artificial Intelligence
- AG Medical Data Science
- AG Medical Deep Learning
- Nachwuchsgruppe Diagnostik und Erforschung von Bewegungsstörungen
- Ehemalige AG Medical Data Engineering
Ansprechpartner
Heinz Handels

Institutsdirektor
Gebäude 64, 2.OG
,
Raum 87
heinz.handels(at)uni-luebeck.de
+49 451 3101 5600
